CDA RhoDb : resp M. Boudvillain CBM
Ressources : Eric Eveno, Yvan Stroppa (Déc 2024 – )
Construction d’une base de connaissances sur la terminaison Rho-dépendante chez les bactéries à l’aide de JBrowse 2
La terminaison Rho-dépendante est un mécanisme clé dans la régulation transcriptionnelle des bactéries, reposant sur l’interaction de la protéine Rho avec l’ARN. Afin de mieux comprendre et caractériser les sites d’interaction potentiels de cette protéine, nous avons mené une approche SELEX (Systematic Evolution of Ligands by EXponential enrichment). Ce travail a permis d’identifier des séquences susceptibles de jouer un rôle crucial dans ce processus biologique.
Pour rendre ces données accessibles et exploitables par la communauté scientifique, nous avons comme objectif de construit une base de connaissances en ligne basée sur JBrowse 2, un navigateur génomique moderne et interactif. Cet outil permet une exploration intuitive des données génomiques et offre une plateforme idéale pour visualiser les résultats de nos analyses.
Objectifs de la base de connaissances :
- Centralisation des données :
- Réunir les séquences identifiées par SELEX en les annotant comme sites potentiels d’interaction de Rho.
- Visualisation intuitive :
- Offrir une représentation claire et interactive des sites d’interaction sur différents génomes bactériens.
- Accessibilité en ligne :
- Fournir un accès web universel, permettant aux chercheurs d’explorer les données sans nécessiter d’installation locale.
- Extension des analyses :
- Intégrer des informations supplémentaires telles que les régions conservées, les motifs spécifiques et les annotations fonctionnelles associées.
Fonctionnalités offertes par JBrowse 2 :
- Exploration des sites identifiés :
- Les séquences enrichies par SELEX sont affichées comme des pistes spécifiques sur les génomes bactériens correspondants.
- Comparaison multi-génomique :
- Les utilisateurs peuvent visualiser la conservation de ces sites entre différentes espèces bactériennes.
- Annotations dynamiques :
- Chaque site est associé à des métadonnées, incluant les scores SELEX, les prédictions fonctionnelles et des données expérimentales complémentaires.
- Compatibilité des formats :
- JBrowse 2 prend en charge des formats standard tels que GFF3, BED, ou FASTA, permettant une intégration transparente de nos résultats.
Perspectives :
Cette base de connaissances a pour ambition de servir de ressource pour :
- Explorer les mécanismes de terminaison transcriptionnelle Rho-dépendante.
- Identifier des cibles potentielles pour des applications biotechnologiques ou thérapeutiques.
- Encourager la collaboration et l’échange d’informations entre chercheurs en génomique bactérienne.
Grâce à JBrowse 2, nous mettons à disposition un outil performant et évolutif, capable d’accompagner les avancées dans ce domaine crucial de la biologie moléculaire.
Procédure d’installation de JBrowse2 sous Linux :
Cette page décrit les différentes opérations à mener pour effectuer cette installation et le déploiement sur une des machines de SPGoO.
Préparation de l’image docker
Se connecter sous le github suivant : https://github.com/biocorecrg/jbrowse_docker/tree/master et récupérer l’ensemble du github via la commande
git clone https://github.com/biocorecrg/jbrowse_docker.gitConstruction de l’image à l’aide de la commande
docker build -t jbrowse2 .Contenu du Dockerfile
# from node image
FROM node:16-buster
LABEL org.opencontainers.image.authors=”toni.hermoso@crg.eu”
ARG JBROWSE_VERSION=1.7.9
ARG SAMTOOLS_VERSION=1.15.1
ARG HTSLIB_VERSION=1.15.1
# Handle dependencies
RUN apt-get update && apt-get -y upgrade && apt-get -y install build-essential git zlib1g-dev \
genometools && \
apt-get clean && echo -n > /var/lib/apt/extended_states
RUN mkdir -p /soft/bin
RUN wget -q https://github.com/samtools/samtools/releases/download/${SAMTOOLS_VERSION}/samtools-${SAMTOOLS_VERSION}.tar.bz2 && \
tar jxf samtools-${SAMTOOLS_VERSION}.tar.bz2 && \
cd samtools-${SAMTOOLS_VERSION} && \
make prefix=/soft/samtools install && \
cd /soft/bin && ln -s /soft/samtools/bin/* . && cd /soft && \
rm -rf *tar.bz2
RUN wget -q https://github.com/samtools/htslib/releases/download/${HTSLIB_VERSION}/htslib-${HTSLIB_VERSION}.tar.bz2 && \
tar jxf htslib-${HTSLIB_VERSION}.tar.bz2 && \
cd htslib-${HTSLIB_VERSION} && \
make prefix=/soft/htslib install && \
cd /soft/bin && ln -s /soft/htslib/bin/* . && cd /soft && \
rm -rf *tar.bz2
# PATH
ENV PATH $PATH:/soft/bin
RUN mkdir -p /srv
WORKDIR /srv
COPY index.js .
COPY package.json .
RUN npm install -g forever
RUN npm install -g @jbrowse/cli@${JBROWSE_VERSION}
RUN npm install
# Volumes
VOLUME /var/www
VOLUME /data
EXPOSE 8080
CMD NODE_ENV=production forever index.js
Une fois le script de création de l’image terminé, vous devez visualiser dans votre dépôt local l’image à l’aide de la commande
docker images
ubuntu@vps:~/CBM/jbrowse_docker$ docker images
REPOSITORY TAG IMAGE ID CREATED SIZE
jbrowse2 latest 94fb0b13e9c1 16 hours ago 1.38GB
Pré-requis: création des répertoires pour les deux volumes
mkdir www && mkdir data Démarrage de l’image
docker run -d --user $(id -u):$(id -g) --name mybrowser -v /my/var/www:/var/www -v /my/data:/data -p 8081:8080 jbrowse2
Paramétrage de l’instance :
Installation des données issues d’Ecoli
docker exec mybrowser samtools faidx /data/GCF_000005845.2_ASM584v2_genomic.fna
docker exec mybrowser bash -c 'jbrowse add-assembly /data/GCF_000005845.2_ASM584v2_genomic.fna --out /var/www/ecoli --load symlink'
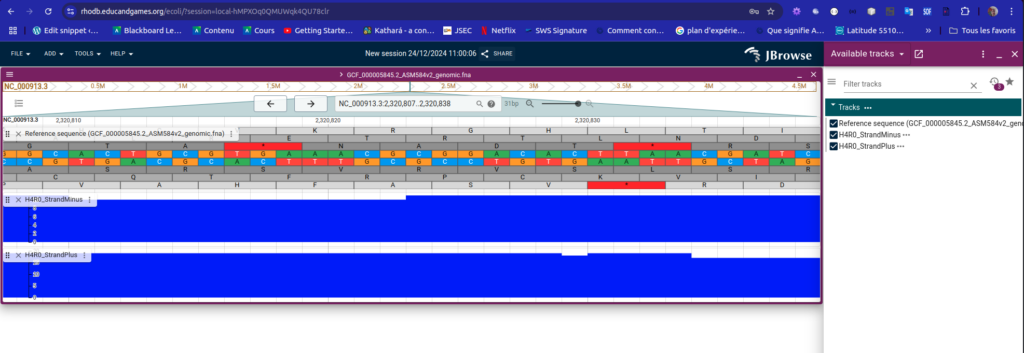
docker exec mybrowser bash -c 'jbrowse add-track /data/H4R0_StrandMinus.bw --name H4R0_StrandMinus --out /var/www/ecoli --load symlink'
docker exec mybrowser bash -c 'jbrowse add-track /data/H4R0_StrandPlus.bw --name H4R0_StrandPlus --out /var/www/ecoli --load symlink'
docker exec mybrowser bash -c 'jbrowse text-index --out /var/www/ecoli'Sécurisation
Modification des paramètres du reverse proxy pour prendre en compte une authentification simple pour le moment.
Exploitation :
Accès via l’url https://rhodb.educandgames.org/ecoli
(12/02/2025 neutralisé en attendant l’hébergement CBM)
Intégration dans Jupyter NoteBook
Comment utiliser JBrowse2 à partir de Jupyter Notebook.
voir doc : https://pmc.ncbi.nlm.nih.gov/articles/PMC9887080/
Extension :
A prévoir une authentification via annuaire LDAP (intégration dans nginx)
Voir https://jbrowse.org/docs/authentication.html

