CDA Aizynthfinder
Ressources SPGoO : Pascal Krezel, Yvan Stroppa (déc 2024 – janv 2025)
Mise en oeuvre d’un service autour de la solution de rétro-synthèse AizynthFinder.
Plusieurs étapes dans cette mise en place.
- La première étape permettra de vérifier l’exhaustivité des éléments fournis sous le github, de consolider l’ensemble, de l’installer sur une machine, de l’exécuter et de vérifier les résultats à partir de molécules connues et de l’expertise des collègues.
- La deuxième étape consistera à encapsuler l’ensemble dans une image Docker, afin de pouvoir le mettre à disposition et le transporter d’une machine à une autre sans perte de temps.
- La troisième étape consistera à travailler sur l’ajout à cet ensemble d’un service Web, qui devra permettre d’invoquer la résolution d’une rétro-synthèse via un appel de type API-REST. Objectif est de pouvoir connecter ce dispositif à un frontal web ou à une application tierce.
- La quatrième étape consistera au développement d’un frontal Web à destination des collègues chimistes qui devrait leur permettre d’interroger via un environnement Graphique ce web service pour un ou plusieurs smiles et de visualiser les résultats de ces rétro-synthèse pour leur molécules.
- La cinquième étape consistera à ajouter la possibilité de compléter la base de connaissance de ce dispositif pour rajouter des réactions/réactifs supplémentaires.
Etape 1 : Installation et vérification :
Fichiers de données et modèles :
A partir des sources déposées sur le github suivant, on installe anaconda et le paramétrage de l’environnement conda comme indiqué dans le fichier README. Ce que ne fournit par le github c’est les fichiers hdf5 contenant les données nécessaires pour exécuter le code fourni et le fichier de configuration nécessaire pour la résolution: config.yml.
A partir de la documentation du site de https://molecularai.github.io/aizynthfinder/ on reconstitue le premier exemple fourni de fichier de configuration:
expansion:
full:
- uspto_expansion.onnx
- uspto_templates.csv.gz
stock:
zinc: zinc_stock.hdf5
Il faut retrouver les fichiers qui ne sont pas présents sur le github des sources. On arrive à retrouver les fichiers sur des sites différents :
- A trained Keras expansion model that is called uspto_expansion.onnx : voir
- A library of unique templates called uspto_templates.csv.gz –> Pascal Krezel
- A stock file in HDF5 format, called zinc_stock.hdf5
Description du contenu de ces trois fichiers :
Contenu de l’archive : “zinc_stock_17_04_20.hdf5
show_hdf5_tree(“zinc_stock_17_04_20.hdf5”)
└── table
├── axis0 (1)
├── axis1 (17422831)
├── block0_items (1)
└── block0_values (1)
Structure et contenu du fichier uspto_templates.csv.gz
template_code retro_template template_hash classification library_occurence
0 [#7;a:4]:[c:3]:c;H0;D3;+0:1:[c:2]>>Br-c;H0;D3;+0:1:[c:3]:[#7;a:4].[IH;D0;+0:5] 0001ead678cb363e3389b34d33a6bd38acc9f88da5403e820b7bc8c2800eb53a 0.0 Unrecognized 19
1 [N;D1;H0:4]#[C:3]-[CH2;D2;+0:1]-[c:2]>>C-C(-C)(-C)-O-C(=O)-CH;D3;+0:1-[C:3]#[N;D1;H0:4] 0002f97a4d38f973095fc7d64488966c9d7b321beb0ea6c38dd61aefac810c0f 0.0 Unrecognized 7
2 [C:3]=[C:2]-[CH2;D2;+0:1]-[S;H0;D2;+0:4]-[c;H0;D3;+0:5]1:[#16;a:6]:[c:7]:[#7;a:8]:[n;H0;D2;+0:9]:1>>C-C(=O)-O-[CH2;D2;+0:1]-[C:2]=[C:3].[S;H0;D1;+0:4]=[c;H0;D3;+0:5]1:[#16;a:6]:[c:7]:[#7;a:8]:[nH;D2;+0:9]:1 0005f9ceb67426e8a8b4a22b0254d8a1bdefd2b2a0a233c966f8b0f67c86738a 0.0 Unrecognized 4
....
....
42552 [#7:4]-C:5-[c:7]1:[c:8]:[c:9]:[c:10]:c:11:[c:13]:1>>F-c;H0;D3;+0:1:[c:3].[#7:4]-C:5-[c:7]1:[c:8]:[c:9]:[c:10]:c:11:[c:13]:1 ffff06aeed0e85fda8980c3e4e178ef6afbda0820acfe7a9558fbaffedeb64d6 0.0 Unrecognized 43
42553 [#7;a:2]:[nH;D2;+0:1]:[c:3]>>C-O-C(=O)-n;H0;D3;+0:1:[c:3] fffff6d4a752349dce0db6afa525867fde3c248d45809392d62549ec926d30d0 0.0 Unrecognized 84Constitution du fichier smiles.txt
Cc1cccc(c1N(CC(=O)Nc2ccc(cc2)c3ncon3)C(=O)C4CCS(=O)(=O)CC4)C
Pour exécuter le script il suffit de lancer la commande suivante :
(aizynth-envv2) ystroppa@dragon:~/ICOA/aizynthfinder_v2/aizynthfinder$ aizynthcli --config config.yml --smiles smiles.txt
Loading template-based expansion policy model from uspto_model.onnx to full
Loading templates from uspto_templates.csv.gz to full
Loading stock from InMemoryInchiKeyQuery to zinc
Selected as stock: zinc
Compounds in stock: 17422831
Selected as expansion policy: full
Done with Cc1cccc(c1N(CC(=O)Nc2ccc(cc2)c3ncon3)C(=O)C4CCS(=O)(=O)CC4)C in 24.1 s and is solved
Output saved to output.json.gzAnalyse et visualisation des résultats à partit du fichier output.json.gz
BenechMarks pour valider les résultats obtenus à partir de la solution :
Molécules d’EMOLGINE
Molécules ICOA
Etape 2 : conteneurisation de la solution
L’idée est de réunir l’ensemble des éléments de aizf et d’ajouter tous les fichiers nécessaires à son fonctionnement. Plusieurs tentatives ont été exécutées avec entre autre anaconda3 mais dans ce cas on a eu un problème de chargement de contexte conda pour exécuter le pip nécessaire “RUN conda activate aizynth-env && python -m pip install aizynthfinder[all]” , erreur de la création d’image lors du chargement du contexte conda. Le système nous indique qu’il faut exécuter au préalable conda init. Du coup on s’est reporté sur miniconda en version 22.11.1 avec en natif un python 3.10 ce qui évite le chargeent de l’environnement conda car on l’a en natif.
Fichier Dockerfile
FROM continuumio/miniconda3:22.11.1
RUN addgroup --gid 1000 icoa
RUN adduser --uid 1000 --gid 1000 --disabled-password --gecos "" icoa
WORKDIR /home/icoa
USER icoa
RUN git clone https://github.com/MolecularAI/aizynthfinder.git
COPY --chown=icoa:icoa config.yml ./
COPY --chown=icoa:icoa smiles.txt ./
COPY --chown=icoa:icoa uspto_model.onnx ./
COPY --chown=icoa:icoa uspto_templates.csv.gz ./
COPY --chown=icoa:icoa zinc_stock_17_04_20.hdf5 ./
ENV bashrc /home/icoa/.bashrc
RUN echo 'export PATH=/home/icoa/.local/bin:$PATH'>> ~/.bashrc
RUN python -m pip install aizynthfinder[all]
Utilisation de l’image :
#docker run -it icoa bash
Syntaxe de la commande aizynthcli :
usage: aizynthcli [-h] --smiles SMILES --config CONFIG [--policy POLICY [POLICY …]] [--filter FILTER [FILTER …]]
[--stocks STOCKS [STOCKS …]] [--output OUTPUT] [--log_to_file] [--nproc NPROC] [--cluster]
[--route_distance_model ROUTE_DISTANCE_MODEL] [--post_processing POST_PROCESSING [POST_PROCESSING …]]
[--pre_processing PRE_PROCESSING] [--checkpoint CHECKPOINT]
Exemple d’exécution dans le conteneur :
icoa@599068b1cb84:~$ aizynthcli --config config.yml --smiles smiles.txt
Loading template-based expansion policy model from uspto_model.onnx to full
Loading templates from uspto_templates.csv.gz to full
Loading stock from InMemoryInchiKeyQuery to zinc
Selected as stock: zinc
Compounds in stock: 17422831
Selected as expansion policy: full
Done with c1(F)cccc(F)c1S(=O)(=O)N(c2c(F)c(c3nc(CC(=O)NC)sc(c4ccnc(Nc5ccccc5)n4)3)ccc2) in 20.1 s and is not solved
Output saved to output.json.gz
Possibilité de démarrer un Jupyter Notebook à partir d’un conteneur via la commande suivante :
docker run -it -p 8888:8888 icoa .local/bin/jupyter notebook --ip='*' --port=8888 --allow-rootIl suffit de relever dans les logs du démarrage du conteneur le token inscrit dans l’url suivante : http://127.0.0.1:8888/?token=865bc85149cfc0274c6ff4f72fb9c2006f41bf547ddeef58 et de lancer un navigateur sur la machine exécutant le conteneur. Ce qui permet d’accéder à jupyter notebook.
Reprendre la doc fournie par aizynthfinder https://molecularai.github.io/aizynthfinder/gui.html pour utiliser en mode graphique les fonctionnalités disponibles. Attention, lors de l’utilisation de l’interface le mode GUI ne fonctionne pas correctement.
Production des images à partir des résultats
Script fournie par AiZF pour l’exploitation des résultats et la production des images
import pandas as pd
from aizynthfinder.reactiontree import ReactionTree
data = pd.read_json("output.json.gz", orient="table")
all_trees = data.trees.values # This contains a list of all the trees for all the compounds
trees_for_first_target = all_trees[0]
for itree, tree in enumerate(trees_for_first_target):
imagefile = f"route{itree:03d}.png"
ReactionTree.from_dict(tree).to_image().save(imagefile)Usage en mode sécurisé :
Pour un usage en contexte sécurisé, nous conseillons de télécharger l’image docker fournie sur le site …. et d’exécuter un conteneur sur cette image pour un usage strictement local.
Etape 3 : aménagement de la solution avec un Web service sous FastAPI : 13/01/2026
Monter un service web sous Python pour permettre des appels en passant le smiles et les paramètres pour obtenir les différents résultats sous forme d’image. Pour ce faire nous installer l’environnement suivant :
| Répertoires ou fichiers | |
| /base | Répertoire avec l’ensemble des fichiers modèles et de data |
| /aizynthfinder | Répertoire avec l’ensemble des scripts du calcul de la rétrosynthèse |
| aizynthYS.py | les chemins d’accès aux différents services |
| config.yml | Le fichier de configuration inquant la localisation des fichiers modèle et data |
| /database | le module de connexion et interrogation de la base de données MongoDB |
| main.py | fichier de démarrage |
| /models | les modèles utilisés dans mes échanges avec le web service |
| /tmp | Répertoire de dépôt des résultats |
| .env | Le fichier de déclaration des variables d’environnement base de données et sécurité |
Dans le répertoire base :
- le fichier modèle onnx : uspto_model.onnx
- le fichier uspto_templates.csv.gz
- le fichier zinc_stock_17_04_20.hdf5
Les adaptations effectuées dans les scripts de base ne concerne que le fichier interfaces/aizynthcli.py qui s’est transformé de la manière suivante : la fonction main a été éclatée en deux parties une fonction load_scartek et une fonction main_scartek. Ceci permet de séparer ces deux actions à des moments différents : le load_scartek sera exécuté par le web service au démarrage et le main_scartek va permettre d’exécuter le traitement de rétrosynthèse. Ce découpage permet de charger la base et le modèle une seule fois et de l’utiliser pour chaque évaluation de rétrosynthèse.
Ce qui nous donne pour les deux méthodes :
# On adapte l'appel de cette fonction au contexte Web service
def load_scartek(Config: str) -> None:
"""Entry point for the aizynthcli command modifie"""
global args, finder, post_processing,pre_processing
args.config=Config
print("chargement du load -- modèle chargé")
finder = AiZynthFinder(configfile=Config)
_select_stocks(finder, args)
post_processing = _load_postprocessing_jobs(args.post_processing)
pre_processing = _load_preprocessing_job(args.pre_processing)
finder.expansion_policy.select(args.policy or finder.expansion_policy.items[0])
finder.filter_policy.select_all()
Et pour la deuxième fonction :
# On adapte l'appel de cette fonction au contexte Web service
def main_scartek(Smiles:str, Output: str) -> None:
"""Entry point for the aizynthcli command modifie"""
global finder , post_processing,pre_processing
argsL=copy.copy(args)
argsL.smiles=Smiles
print("traitement" + argsL.smiles+ " " + Output)
params = [
Smiles,
finder,
Output,
argsL.cluster,
argsL.route_distance_model,
post_processing,
pre_processing,
argsL.checkpoint,
]
_process_multi_smiles(*params)
Intégration de ces deux points d’entrée dans notre dispositif de web service :
Le main.py va nous permettre de charger les différents modules dédiés à FastAPI, MongoDB et à initialiser les accès base et le chargement du modèle pour aizynthfinder. Ensuite on rajoute les différents points d’entrée de notre web service.
app = FastAPI()
app.include_router(aizynthfinder_router)
settings=Settings()
@app.on_event("startup")
async def chargement_model():
load_scartek("./config.yml")
await settings.initialize_database()
Définition des points d’entrée : aizynthfinder_router pour déclencher les différents traitements. On a besoin de deux entrées pour produire la rétrosynthèse et ensuite pour produire les images associées. Pour chaque fonction on vérifiera au préalable si la rétrosynthèse a déjà été effectuée. Si c’est le cas on indiquera le code spécifique pour l’accès aux images.
requirements.txt
absl-py==2.1.0
aiohappyeyeballs==2.4.4
aiohttp==3.11.11
aiosignal==1.3.2
alabaster==1.0.0
alembic==1.14.0
annotated-doc==0.0.4
annotated-types==0.7.0
anyio==4.7.0
apted==1.0.3
argon2-cffi==23.1.0
argon2-cffi-bindings==21.2.0
arrow==1.3.0
asttokens==3.0.0
async-lru==2.0.4
async-timeout==5.0.1
attrs==24.3.0
autopage==0.5.2
babel==2.16.0
bcrypt==5.0.0
beanie==2.0.1
beautifulsoup4==4.12.3
black==24.10.0
bleach==6.2.0
blosc2==2.7.1
certifi==2024.12.14
cffi==1.17.1
charset-normalizer==3.4.1
click==8.1.8
cliff==4.8.0
cmaes==0.11.1
cmd2==2.5.8
coloredlogs==15.0.1
colorlog==6.9.0
comm==0.2.2
commonmark==0.9.1
contourpy==1.3.1
cycler==0.12.1
debugpy==1.8.11
decorator==5.1.1
defusedxml==0.7.1
Deprecated==1.2.15
dnspython==2.8.0
docutils==0.21.2
ecdsa==0.19.1
entrypoints==0.4
exceptiongroup==1.2.2
executing==2.1.0
fastapi==0.128.0
fastjsonschema==2.21.1
flake8==7.1.1
flatbuffers==24.12.23
fonttools==4.55.3
fqdn==1.5.1
frozenlist==1.5.0
fsspec==2024.12.0
greenlet==3.1.1
grpcio==1.68.1
h11==0.14.0
httpcore==1.0.7
httpx==0.28.1
humanfriendly==10.0
idna==3.10
imagesize==1.4.1
importlib_resources==6.4.5
iniconfig==2.0.0
ipykernel==6.29.5
ipython==8.31.0
ipython-genutils==0.2.0
ipywidgets==7.8.5
isoduration==20.11.0
jedi==0.19.2
Jinja2==3.1.5
joblib==1.4.2
json5==0.10.0
jsonpointer==3.0.0
jsonschema==4.23.0
jsonschema-specifications==2024.10.1
jupyter==1.1.1
jupyter-console==6.6.3
jupyter-events==0.11.0
jupyter-lsp==2.2.5
jupyter_client==7.4.9
jupyter_core==5.7.2
jupyter_server==2.15.0
jupyter_server_terminals==0.5.3
jupyterlab==4.3.4
jupyterlab_pygments==0.3.0
jupyterlab_server==2.27.3
jupyterlab_widgets==1.1.11
jupytext==1.16.6
kiwisolver==1.4.8
lazy-model==0.4.0
lightning-utilities==0.11.9
llvmlite==0.43.0
Mako==1.3.8
Markdown==3.7
markdown-it-py==3.0.0
MarkupSafe==3.0.2
matplotlib==3.10.0
matplotlib-inline==0.1.7
mccabe==0.7.0
mdit-py-plugins==0.4.2
mdurl==0.1.2
mistune==3.1.0
molbloom==2.2.1
motor==3.7.1
mpmath==1.3.0
msgpack==1.1.0
multidict==6.1.0
mypy==1.14.0
mypy-extensions==1.0.0
myst-parser==4.0.0
nbclassic==1.1.0
nbclient==0.10.2
nbconvert==7.16.4
nbformat==5.10.4
nbsphinx==0.9.6
ndindex==1.9.2
nest-asyncio==1.6.0
netron==8.0.8
networkx==2.8.8
notebook==6.5.7
notebook_shim==0.2.4
numba==0.60.0
numexpr==2.10.0
numpy==1.26.4
numpydoc==1.8.0
nvidia-cublas-cu11==11.10.3.66
nvidia-cuda-nvrtc-cu11==11.7.99
nvidia-cuda-runtime-cu11==11.7.99
nvidia-cudnn-cu11==8.5.0.96
onnxruntime==1.23.2
optuna==2.10.1
overrides==7.7.0
packaging==24.2
pandas==1.5.3
pandocfilters==1.5.1
paretoset==1.2.4
parso==0.8.4
passlib==1.7.4
pathspec==0.12.1
pbr==6.1.0
pep8==1.7.1
pexpect==4.9.0
Pillow==9.5.0
platformdirs==4.3.6
pluggy==1.5.0
prettytable==3.12.0
prometheus_client==0.21.1
prompt_toolkit==3.0.48
propcache==0.2.1
protobuf==3.20.1
psutil==6.1.1
ptyprocess==0.7.0
pure_eval==0.2.3
py-cpuinfo==9.0.0
pyasn1==0.6.1
pycodestyle==2.12.1
pycparser==2.22
pydantic==2.10.4
pydantic-settings==2.12.0
pydantic_core==2.27.2
pyDeprecate==0.3.2
pyflakes==3.2.0
Pygments==2.18.0
pymongo==4.16.0
pyparsing==3.2.0
pyperclip==1.9.0
pytest==8.3.4
python-dateutil==2.9.0.post0
python-dotenv==1.2.1
python-jose==3.5.0
python-json-logger==3.2.1
pytorch-lightning==1.6.5.post0
pytorch-tree-lstm==0.1.3
pytz==2024.2
PyYAML==6.0.2
pyzmq==26.2.0
rdchiral==1.1.0
rdkit==2022.9.5
recommonmark==0.7.1
referencing==0.35.1
requests==2.32.3
rfc3339-validator==0.1.4
rfc3986-validator==0.1.1
rpds-py==0.22.3
rsa==4.9.1
scikit-learn==1.6.0
scipy==1.14.1
seaborn==0.13.2
Send2Trash==1.8.3
six==1.17.0
sniffio==1.3.1
snowballstemmer==2.2.0
soupsieve==2.6
Sphinx==8.1.3
sphinxcontrib-applehelp==2.0.0
sphinxcontrib-devhelp==2.0.0
sphinxcontrib-htmlhelp==2.1.0
sphinxcontrib-jsmath==1.0.1
sphinxcontrib-qthelp==2.0.0
sphinxcontrib-serializinghtml==2.0.0
SQLAlchemy==2.0.36
stack-data==0.6.3
starlette==0.50.0
stevedore==5.4.0
sympy==1.13.3
tables==3.10.1
tabulate==0.9.0
tensorboard==2.18.0
tensorboard-data-server==0.7.2
terminado==0.18.1
threadpoolctl==3.5.0
timeout-decorator==0.5.0
tinycss2==1.4.0
tomli==2.2.1
torch==1.13.1
torchmetrics==1.5.2
tornado==6.4.2
tqdm==4.67.1
traitlets==5.14.3
types-python-dateutil==2.9.0.20241206
typing-inspection==0.4.2
typing_extensions==4.12.2
uri-template==1.3.0
urllib3==2.3.0
uvicorn==0.40.0
wcwidth==0.2.13
webcolors==24.11.1
webencodings==0.5.1
websocket-client==1.8.0
Werkzeug==3.1.3
widgetsnbextension==3.6.10
wrapt==1.17.0
yarl==1.18.3
Dockerfile
FROM python:3.11
WORKDIR /app
COPY requirements.txt /app
RUN pip install --upgrade pip && pip install -r /app/requirements.txt
RUN pip install onnxruntime
EXPOSE 8005
COPY ./ /app
CMD ["python", "main.py"]
docker-compose.yml
version: "3"
services:
api:
build: .
image: scartekaizf:latest
ports:
- "8005:8005"
env_file:
- .env.prod
database:
image: mongo
ports:
- "27017"
volumes:
- data:/data/db
volumes:
data:
Etape 4 : développement d’une interface web dédiée
Habiller l’ensemble d’un frontal web pour permettre des manipulations simples des utilisateurs.
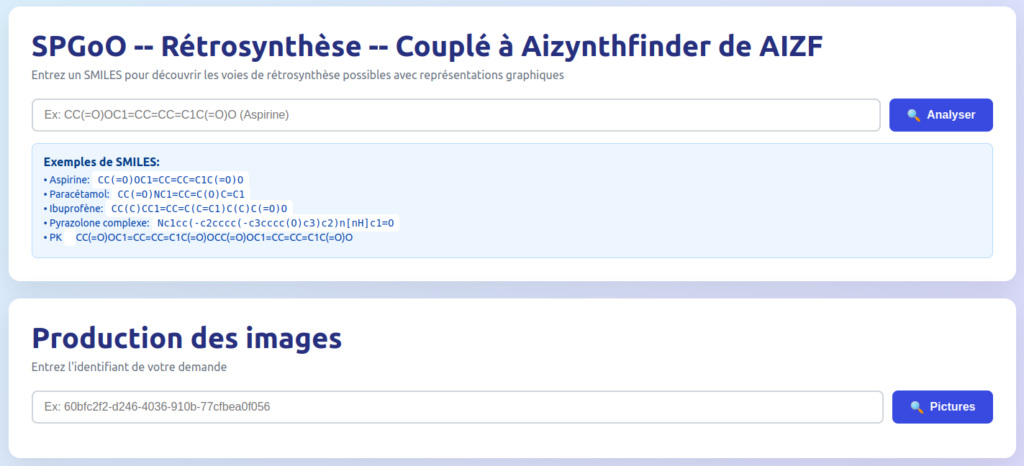
Et l’affichage des résultats après la résolution de la rétrosynthèse
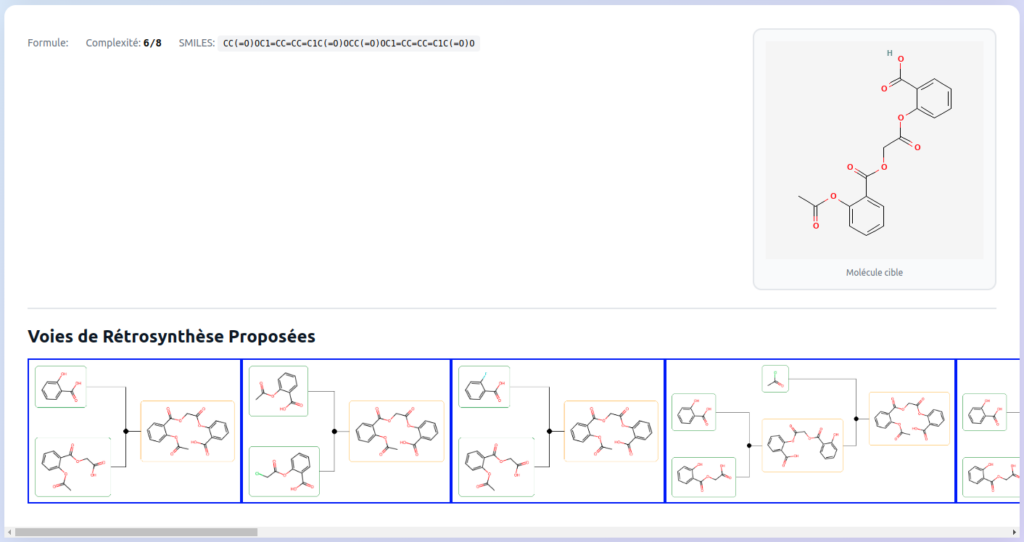
Etape 5 : Extensions possibles
Plusieurs extensions dans ce type de service , la possibilité d’annoter les rétrosynthèse pour avoir une explication plus détaillée sur le procédé, la possibilité de compléter les résultats par des compléments de LLM : de type Ollama ou autre modèle.
Un couplage avec Claude d’Anthropic devrait permettre de compléter les rétrosynthèses, ce couplage peut être réalisé au niveau du smile ou directement au niveau des résultats (images) pour décrire le procédé de rétrosynthèse.
Appel du service Claude d’Anthropic
const response = await fetch('https://api.anthropic.com/v1/messages', {
method: 'POST',
headers: {
'Content-Type': 'application/json',
'x-api-key': apiKey,
'anthropic-version': '2023-06-01'
},
body: JSON.stringify({
model: 'claude-sonnet-4-20250514',
max_tokens: 1024,
messages: conversationHistory
})
});