Neo4J , base de données orientée graphe
Description de la mise en oeuvre de Neo4J dans le cadre du projet emolgine:
Une base Neo4J est constituée de Noeuds de différents types (label) ainsi que de relations de différents type.
Dans la base Neo4J d’emolgine, les types de noeuds définis sont les suivants:
– Molecule (pour défnir les molécules),
– Cluster_mol (pour rassembler les molécules au sein de cluster de molécules similaires),
– Site (pour définir des sites au sein d’une protéine),
– Protein (pour définir des protéines).
Ces noeuds sont reliés entre eux par les relations suivantes:
– FROM: entre Site et Protein,
– CFIT: entre Cluster_mol et Site pour indiquer que les molécules rassemblées au sein d’un cluster de molécules similaires ont de l’affinité potentielle pour un site de protéine donné.
– SMI1: entre Cluster_mol pour indiquer leur similarité,
– SIM2: entre Molecule pour indiquer leur similarité.
En utilisant la fonction “load CSV” du langage cypher, il est possible de construire une base de 2 500 000 de noeuds et 20 000 000 de relations en 20 minutes. L’ objectif est de pouvoir gérér une base contenant au moins plusieurs centaines de millions de molécules avec probablement au moins un milliard de relations.
Le site emolgine permet de lancer des requêtes sur la base Neo4J et de visualiser le résultat sous forme d’un graphe obtenu avec D3.js.
Schéma de la base de données
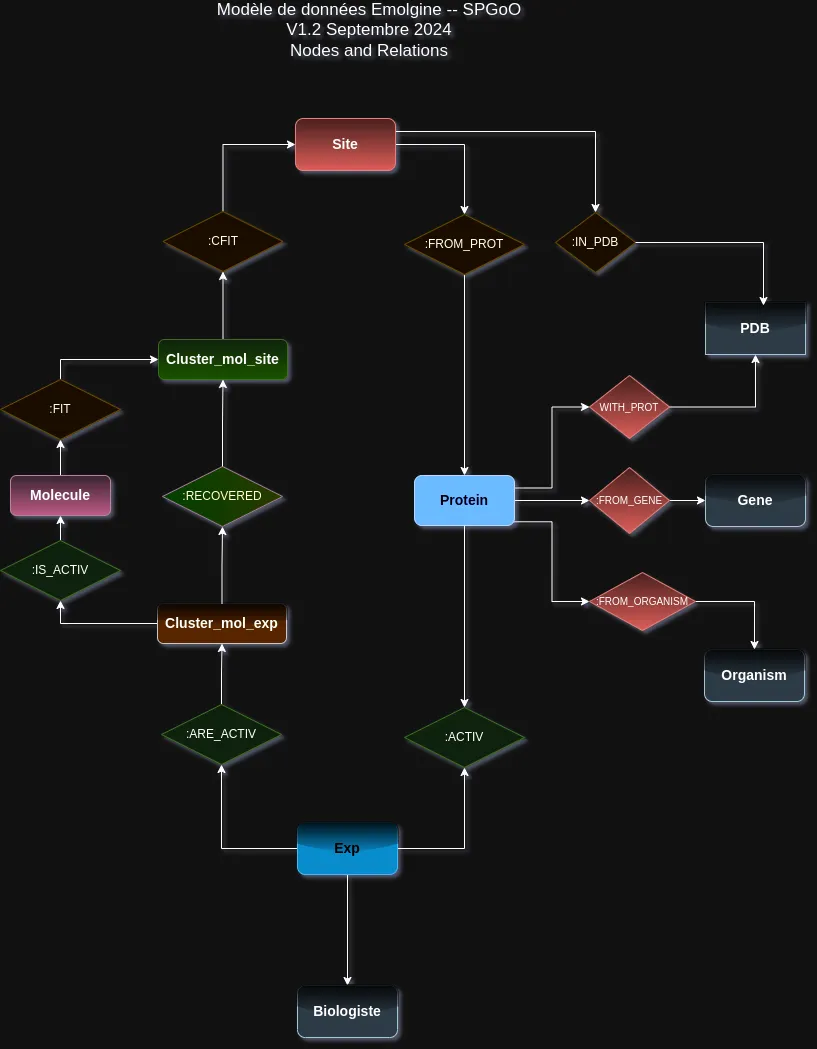
Exemples et explications des requêtes utilisées à partir de ce schéma en Cypher.
Réf : Building Knowledge Graphs – A practioner’s Guide O’Reilly
Caractériser le contenu de la base à partir des relations
- MATCH ()-[r:FIT]->() RETURN count()
- MATCH ()-[r:CFIT]->() RETURN count()
- MATCH ()-[r:FROM]->() RETURN count(*)
Caractériser les attributs associés à la relation FIT
- MATCH ()-[r:FIT]->() RETURN MAX(r.efficiency),MIN(r.efficiency),AVG(r.efficiency)
- MATCH ()-[r:FIT]->() RETURN MAX(r.affinity),MIN(r.affinity),AVG(r.affinity)
Caractériser la répartition en nombre de molécules la relation FIT selon les deux attributs affinity et efficiency
- unwind [0.25,0.386,0.522,0.658,0.794,0.93,1.066,1.202,1.338,1.474,1.61] as efficience unwind range(6,30) as affinity match ()-[r:FIT]-() where affinity<= r.affinity <affinity+1 and efficience<=r.efficiency<efficience+0.136 return affinity,efficience,count(r)
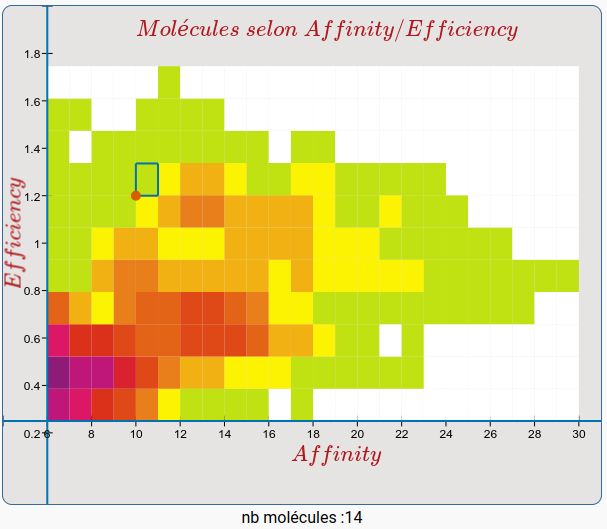
Requêtes avec retour JSON
MATCH (csite:Cluster_mol_site)<-[x:FIT]-(mol:Molecule) RETURN csite.id as Site, collect(mol.id) AS molecules LIMIT 5
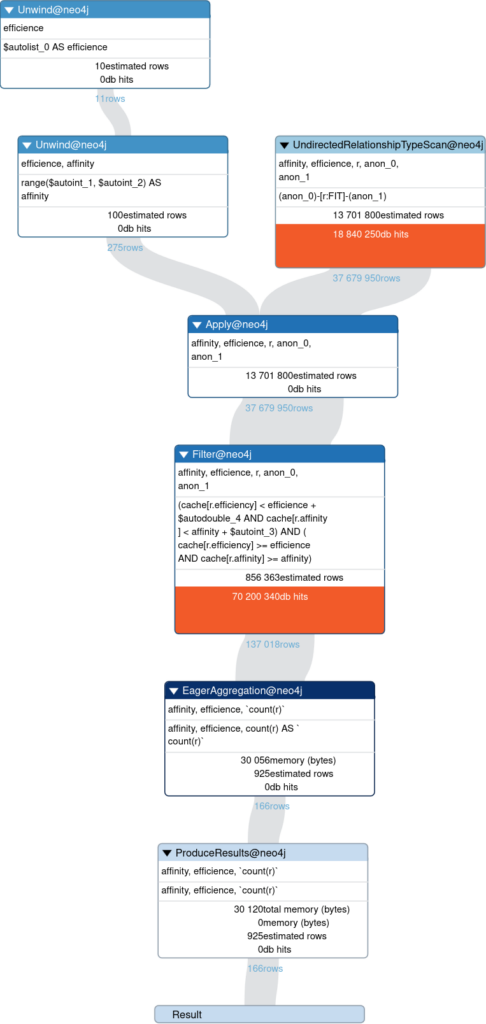
Optimisation de la requête de la répartition
Exécution avec production du plan d’exécution et création d’index
PROFILE unwind [0.25,0.386,0.522,0.658,0.794,0.93,1.066,1.202,1.338,1.474,1.61] as efficience unwind range(6,30) as affinity match ()-[r:FIT]-() where affinity<= r.affinity <affinity+1 and efficience<=r.efficiency<efficience+0.136 return affinity,efficience,count(r)
create index fitrelationaffinity for ()-[r:FIT]-() on r.affinity
create index fitrelationefficiency for ()-[r:FIT]-() on r.efficiency
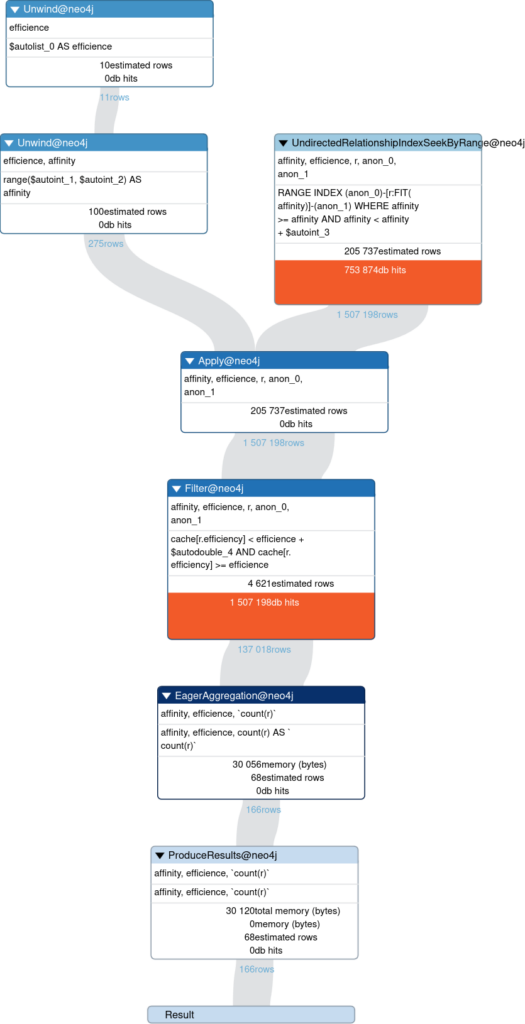
Exécution avec production du plan d’exécution avec les index cette fois-ci.
Explain
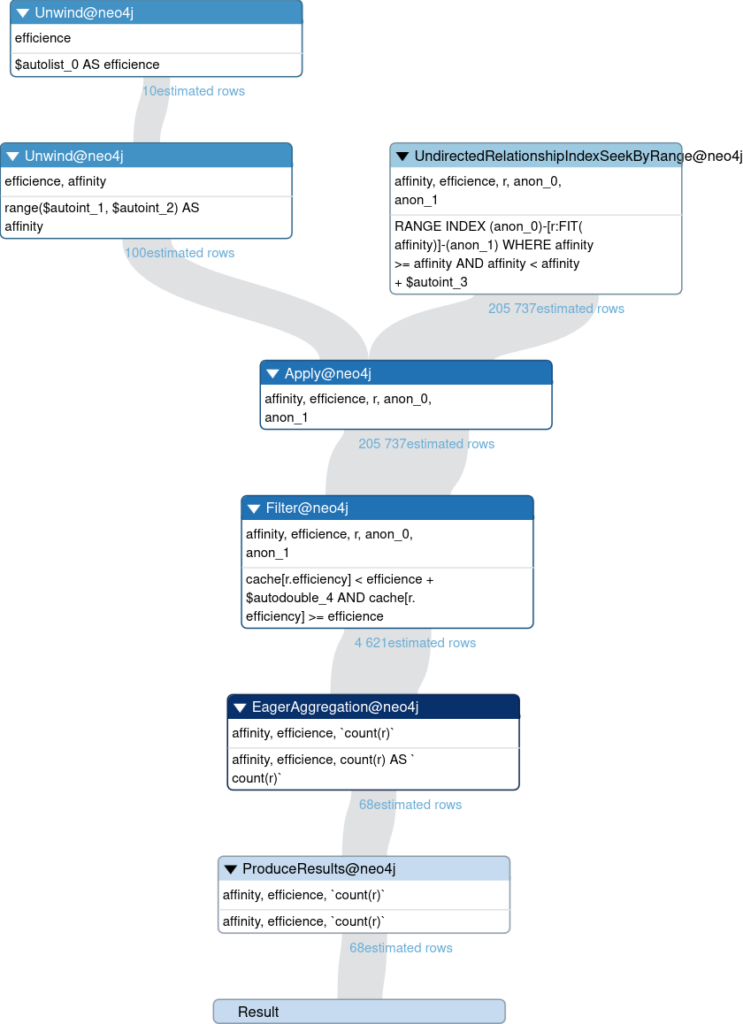
Profile
Quelques instructions utilises
Documentation sur une procédure : call apoc.help(“apoc.export”)
Version d’apoc : return apoc.version() as output;
Export de données:
WITH "requete " AS query CALL apoc.export.csv.query(query,"molecules.csv",{})
YIELD file, source, format, nodes, relationships, properties, time, row, batchsize,batches,done, data
RETURN file, source, format, nodes, relationships, properties, time, row, batchsize,batches,done, dataInstructions classiques
Supprimer tout un ensemble de relation : MATCH ()-[n:import]-() DELETE n
Tout supprimer : MATCH (x) CALL { WITH x DETACH DELETE x }
Documentations :
