CDA EMOLGINE™
Ressources SPGoO : PK, YS, EE (juin 2024 – juin 2025)
Constitution d’une Plateforme de traitement et d’extractions de données autour de solution de type WordPress et de système de base de données de type Graphe Neo4J. Les bibliothèques js utilisées pour les présentations sont jMol, et D3.JS.
Objectif: La plateforme emolgine se veut un outil de chémoinformatique en ligne permettant de rechercher des molécules thérapeutiques pertinentes. Pour le moment, les outils mis en oeuvre sont:
– un générateur de molécules à partir de fragments repositionnés,
– une base de données orientée graphe (Neo4J),
– wordpress pour la création du site,
– javascript pour la programmation du site.
Avec le concours d’étudiants de L3 en informatique à Orléans, une première version d’emolgine a pu être réalisée. Celle-ci utilise une base de données Neo4J contenant :
- Des protéines
- Des clusters de molécules
- Des sites biologiques
- Des molécules
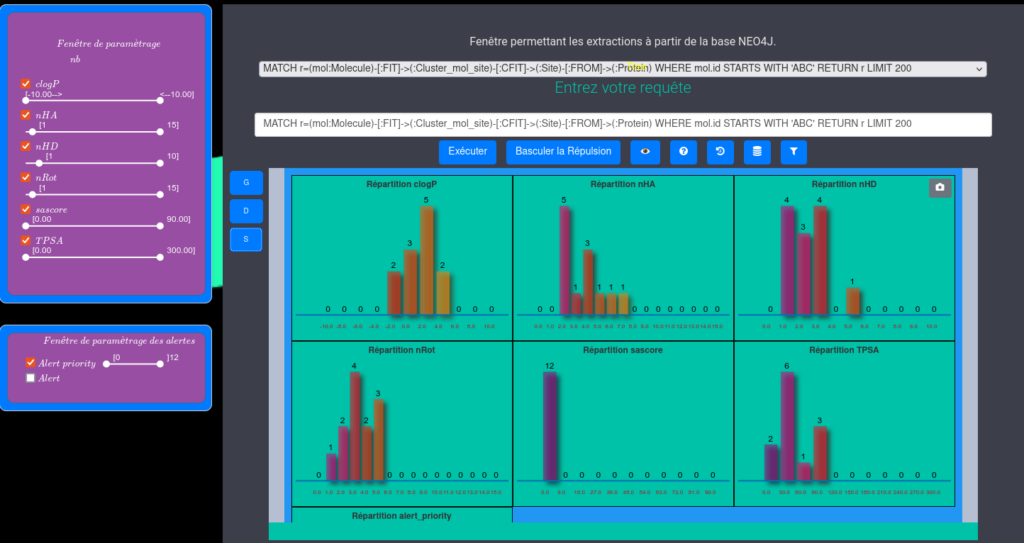
L’utilisateur peut ainsi lancer des requêtes et obtenir un résultat sous la forme d’un graphe grâce au module D3.js.
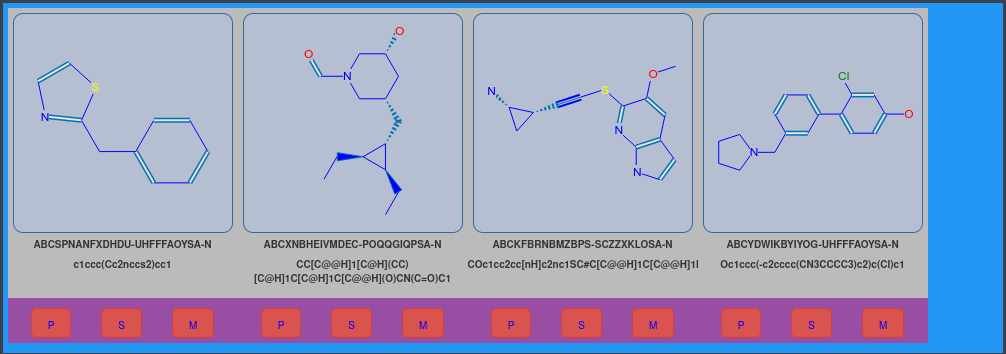
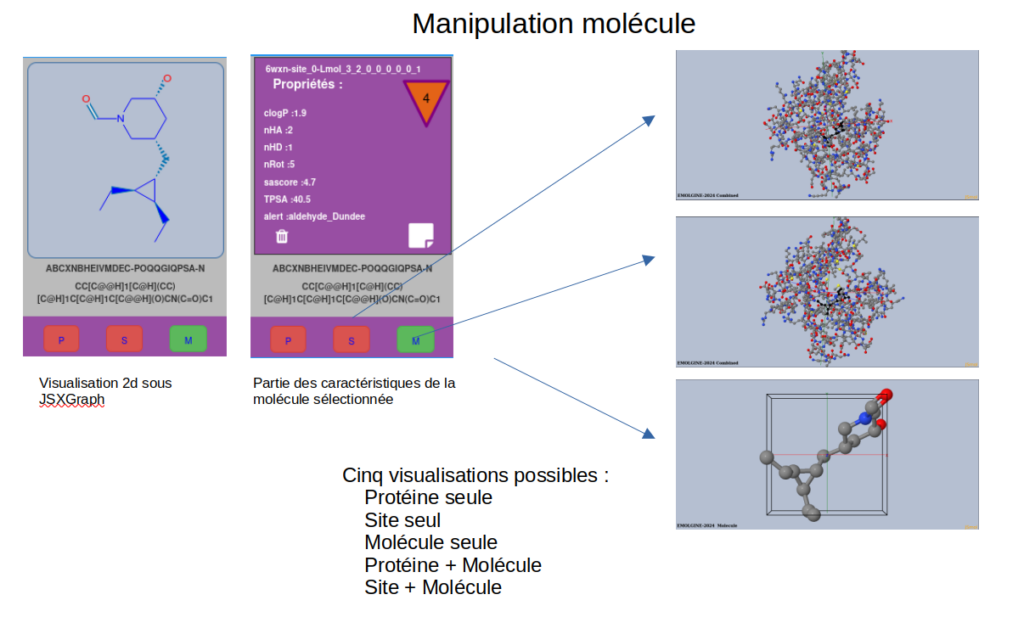
Aussi, en cliquant sur le noeud d’une molécule, l’utilisateur peut la visualiser en 2D et en 3D au sein du site de la protéine où elle a été générée.
Enfin, simplement en cliquant sur le noeud d’une protéine, des informations complémentaires peuvent être obtenues instantanément à l’aide de requêtes envoyées sur les sites RCSB et Uniprot. Cela montre que toute information n’ayant pas été stockée dans la base de données peut être ajoutée rapidement.
Ceci constitue une première étape montrant que les porteurs du projet emolgine maîtrisent désormais les points suivants:
- La création d’une base Neo4J,
- La création d’un site wordpress et sa mise en ligne,
- L’interrogation de la base depuis le site,
- La présentation des résultats sous forme de graphes avec le module 3D.js
- La présentation des molécules en 2D et 3D avecles modules jmol et jsmol.
- La recherche d’information complémentaire quelconque en javascript.
D’autres savoir-faires informatiques vont venir s’ajouter prochainement.
Architecture de la plateforme
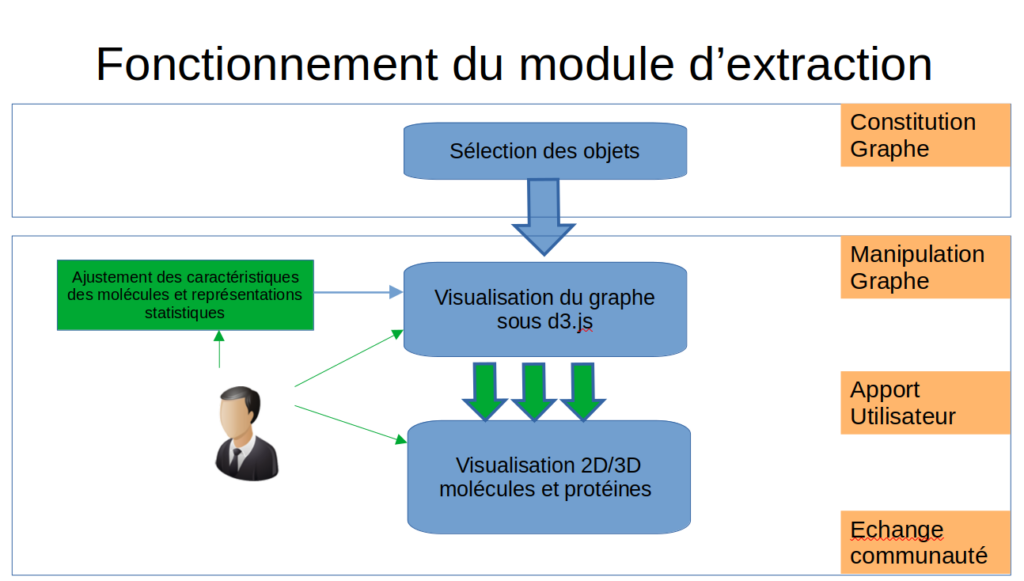
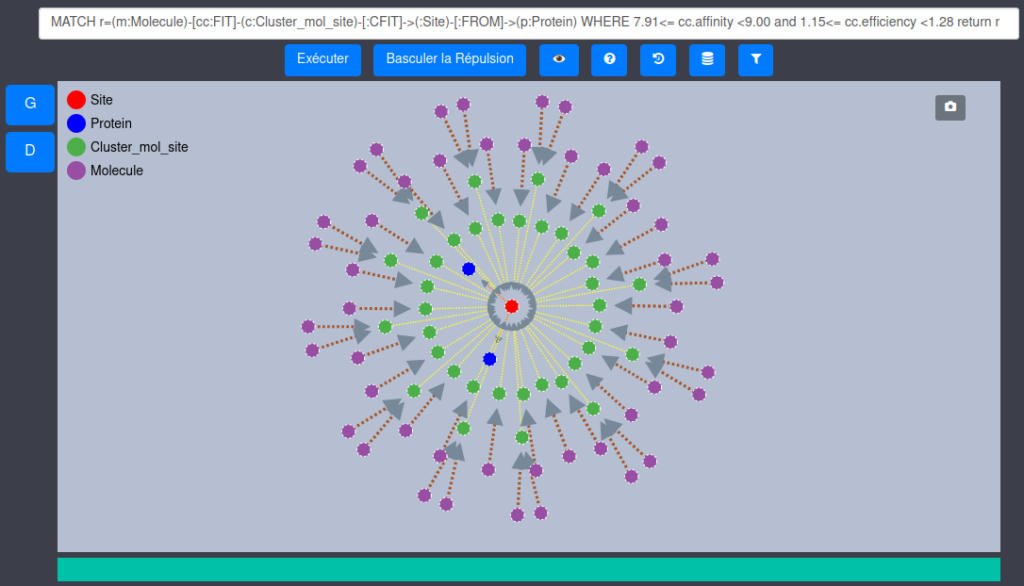
Illustration d’une requête et affichage des résultats sous D3.js
Description des fonctionnalités développées sur le visuel D3.js (à compléter)
Visualisation d’histogrammes associés aux grandeurs caractéristiques des molécules (à compléter).
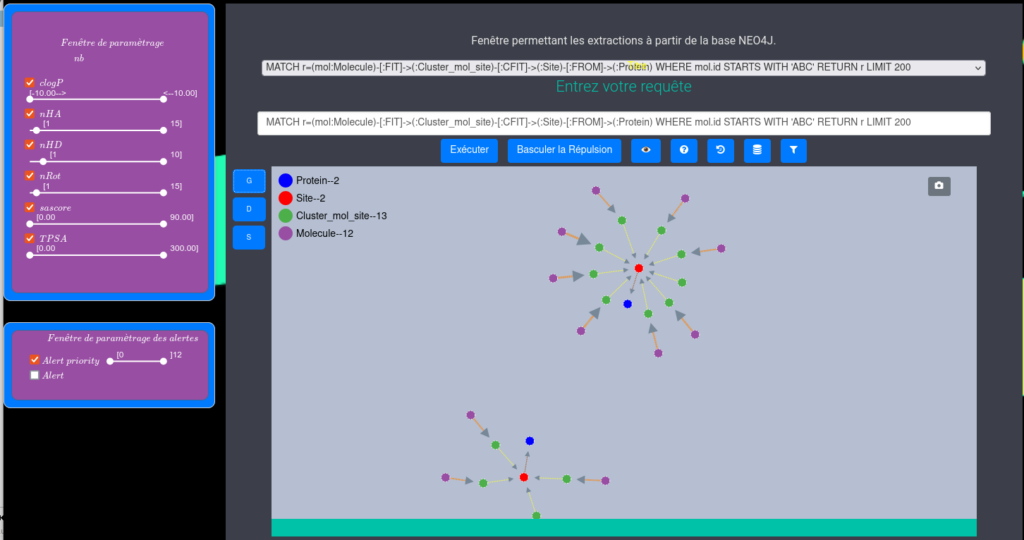
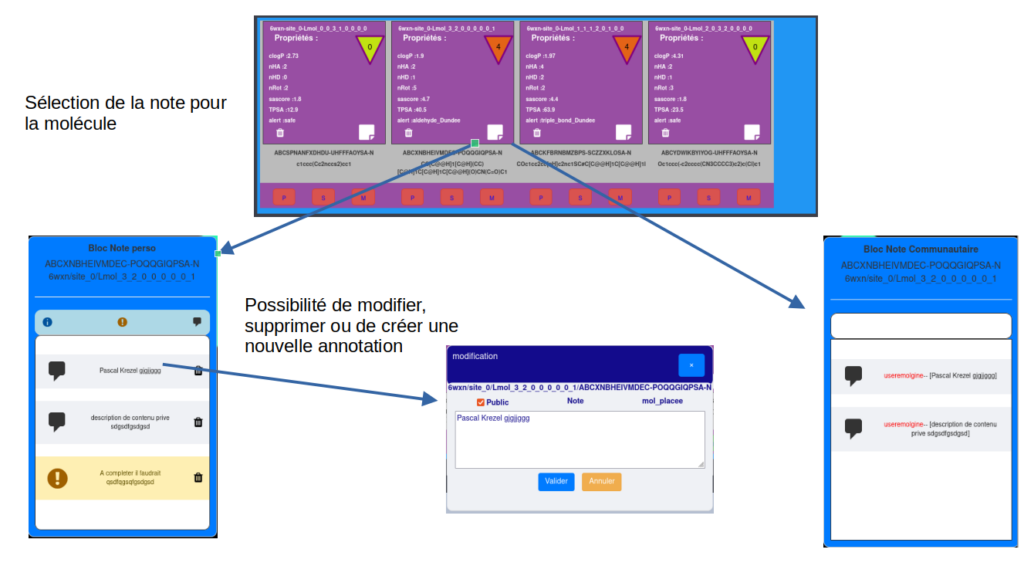
Une fois la sélection des molécules effectuée, on peut consulter les caractéristiques de chacune en cliquant dessus et en visualisant la fiche correspondante à l’aide de JSXGraph. En cliquant sur la structure de la molécule on peut également consulter les propriétés associées et consulter les annotations éventuelles.
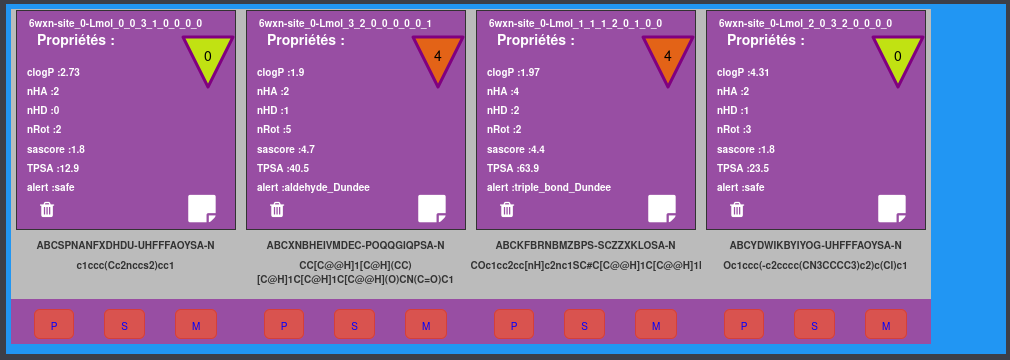
Principe des annotations dans Emolgine (à compléter)
Contrôle et historisation des requêtes (à compléter)
Visualisation sous JSMOL de représentations 3D cumulées.
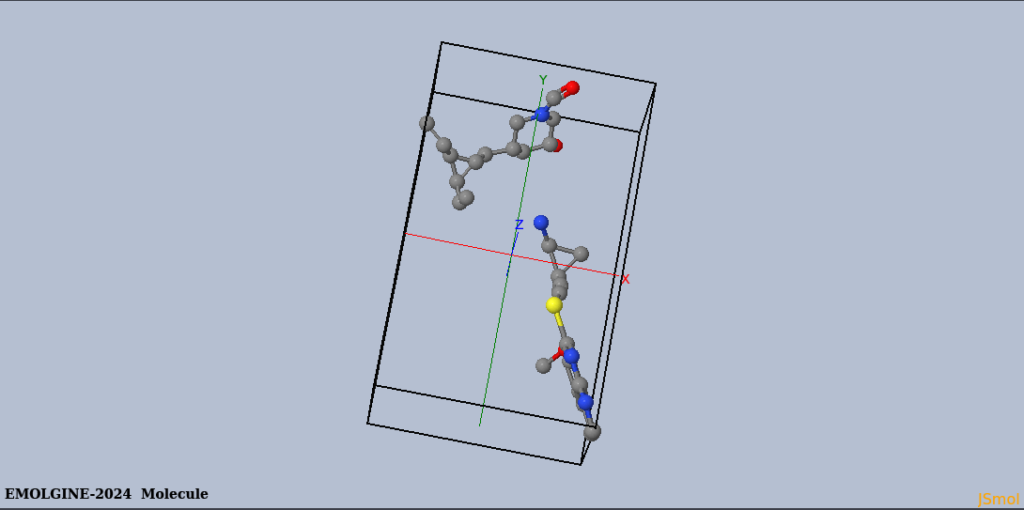
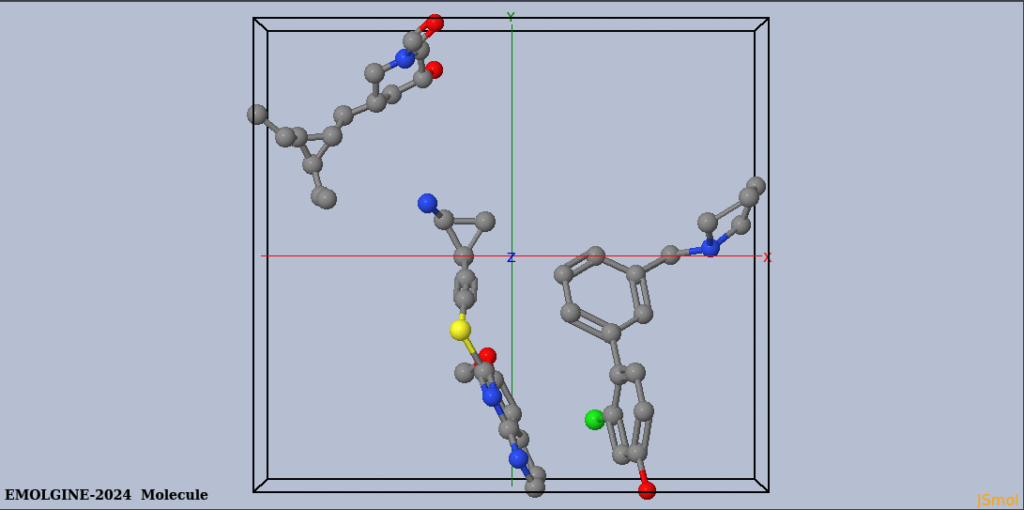
Principe de la fusion des fichiers pdb (à compléter)
On ne peut pas cumuler plusieurs fichiers pdb de molécules dans jmol car ils ont tous la même numérotation des atomes commençant à 1 d’où des liens supplémentaires entre eux qui génèrent une confusion dans l’interprétation. Il est donc nécessaire de décaler cette numérotation. Un script php a été élaboré pour gérer cette situation en prenant du côté serveur le ou les fichiers pdb des molécules à visualiser et en renumérotant tous les atomes et les connexions entre eux à partir de 3000. Chaque fichier cumulé se voit avec une numérotation décalée de 50 ce qui permet de s’assurer qu’il n’y a pas de redondance de numéro et de permettre ensuite le cumul avec les fichiers pdb de la proteine ou du site.
Assistant de requêtage pour la plateforme Emolgine (à compléter)
