RCaWS : Ressources de Calculs accessibles par Web Service
Projet exploratoire d’interfaçage entre plateformes Web et ressources de calculs mutualisées type Cascimodot (cluster sous Slurm).
Objectifs
Dans le cadre de nos développements sur les plateformes Web Emolgine et IAML, nous avons souvent besoin de déporter des traitements “lourds” sur des ressources de calculs adaptées telles que des clusters ou des orchestrateurs. Pour cela, nous souhaitons réalisé ce déport en mode asynchrone sur des files d’attente appropriées. Plusieurs types de problème se posent alors dans cette relation frontal – ressources mutualisées :
- problèmes de sécurité
- problèmes d’échange entre les deux environnements
Nous allons procéder par étapes. Dans un premier temps nous caractériserons les différents problèmes à résoudre et envisagerons ensuite comment procéder pour expérimenter une solution répondant à toutes les exigences. Ensuite nous explorerons l’état de l’art dans ce domaine et regarderont attentivement ce qui se fait du côté des MLOPS et si possible profiter du retour d’expérience des collègues du CC-IN2P3.
Notion de sécurité :
L’accès au frontal se fera via https et avec authentification locale ou par fédération. Cela implique une solution certifiée et acceptée par l’institution.
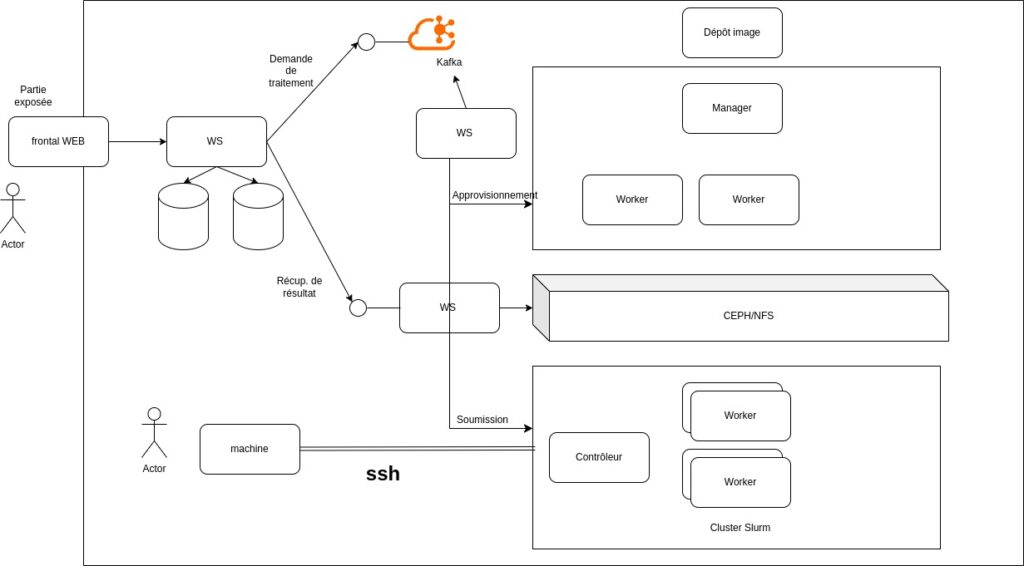
L’accès aux ressources de calculs ne pourra en aucun être fait directement par l’utilisateur. Celui-ci soumettra une requête via l’interface web et la soumission se fera en mode asynchrone via un web service apparié (token) au frontal. C’est le web service qui ira poster dans une liste de soumission la demande de l’utilisateur (kafka ou autre solutions de MOM).
Echange entre les différents contexte :
Demande et Data associées : les échanges entre le frontal et les RC se feront via un web service. L’accès au WS sera conditionné par token (jwt) pour garantir l’exclusivité des accès aux frontaux autorisés. Dans le cas de données volumineuses différentes solutions seront proposées : soit un transfert direct sur un espace approprié associé au WS, soit un dépôt sur un site tiers avec transfert de l’URL et de l’autorisation associé (données sensibles).
Solveurs : les solveurs seront encapsulés dans une image afin de garantir leur intégrité et fournir les dépendances associées. Des circuits de validation de ces images seront à prévoir dans le contexte cible. Les images seront déposées via un circuit interne sur un dépôt certifié, associé aux clusters. Les solveurs peuvent évoluer. le système doit permettre de cumuler plusieurs versions d’un même solveur et de déterminer ceux à utiliser.
Prototypage :
Contexte d’EMOLGINE
Nous souhaitons générer des molécules spécifiques à un site d’une protéine à l’aide d’un algorithme de “growing” et ce à partir de différents fragments. Pour ce faire , l’utilisateur doit pouvoir sélectionner une protéine et définir le site à partir d’une interface web. Après éventuellement la modification de certains paramètres proposés par défaut, il devra pouvoir soumettre un growing sur les ressources calculs. Pour cela, le traitement de growing sera préalablement encapsulé dans une image. Le résultat de ce calcul sera ensuite directement déposé dans un espace dédié pour être ajouté à la base de données Neo4J de la plateforme.
Paramètres à définir à l’aide de l’interface web:
- Les points qui définissent le centre du site
Paramètres par défaut pouvant être modifiés:
- La distance max des points qui définissent le centre du site.
- Le nombre d’itération (le nombre de fois où on va ajouter des fragments aux molécules générées)
- Le nombre max de départ de growing sur une nouvelle molécule (défaut: 2)
- Le nombre max de molécules générées à partir d’une molécule
- La valeur max de l’efficiency pour sélectionner les molécules pouvant passer à l’itération suivante
- Le nombre de fragments utilisés pour faire le growing
- Le nombre de fragments utilisés comme point de départ du growing
- Le nombre max de possibilités de départ à partir d’un atome
Contexte IAML :
Dans le cadre de la plateforme IAML, la problématique sera plus complexe due au volume des données à déplacer pour le traitement. Pour cela des dispositifs classiques de upload dans des structures de base de données de type NOSQL seront utilisés. Pour les traitements, il faudra envisager des encapsulations automatiques des solveurs pour qu’ils soient exécutés suite à une demande de soumission.
Encapsulation :
Deux solutions d’encapsulation seront étudiées Podman et Singularity. Dans ces encapsulations il faudra pouvoir exécuter du code sur CPU ou GPU.
Deux méthodes de mises en oeuvre :
Orchestrateur de type K8S : besoin d’une station de travail avec 4 VMs. Au sein de SPGoO on dispose d’un cluster K8S en version 1.30 disponible ainsi qu’un serveur de dépôt pour les images …
Cluster — Slurm : à mettre en oeuvre.
Architecture de tests de la proposition :