Au sein du centre de recherche d’IBM en Ireland, un projet de passage entre deux représentations de molécules à été réalisé avec en entrée des “fingerprints” de Morgan et en sortie des smiles. Les descripteurs de Morgan indiquent quelles sous-structures sont obtenues en partant de chaque atome et en allant jusqu’à une distance de 2 ( par exemple) de celui-ci. Etant donné la multitude de possibilités, il n’est pas possible de résumer cela simplement et par conséquent ces informations subissent un hashage pour être compressées en un nombre réduit de bits, souvent 2048. En ce qui concerne les smiles, il s’agit de la méthode la plus classique pour coder la structure d’une molécule. Le passage entre ces deux formes de représentation n’est pas évident à cause du hashing.
Le problème lié aux fingerprints, vient du fait qu’ils indiquent la présence ou non d’une sous-structure, mais n’indiquent pas le nombre de fois qu’elle est répétée au sein d’une molécule.
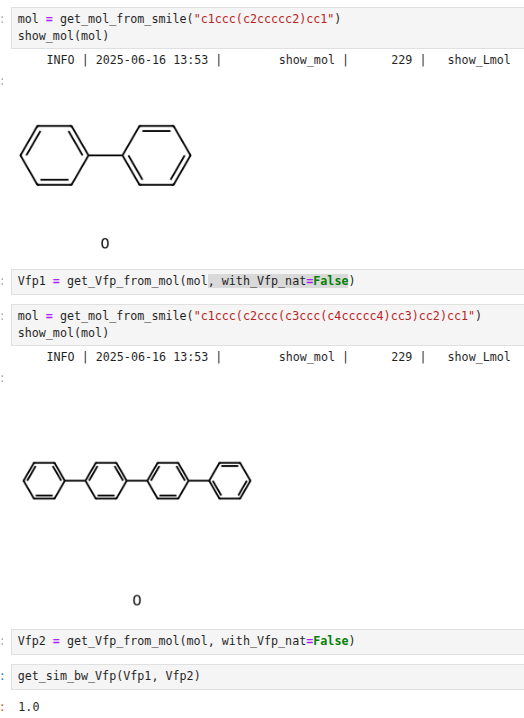
Les deux molécules ci-contre ont le même fingerprint de Morgan pour un rayon de 2.
Ainsi, l’outil développé par l’équipe d’IBM a forcément eu du mal à apprendre combien de fois il doit générer une sous-structure donnée à partir d’un fingerprint de Morgan qui doit correspondre à différents smiles !
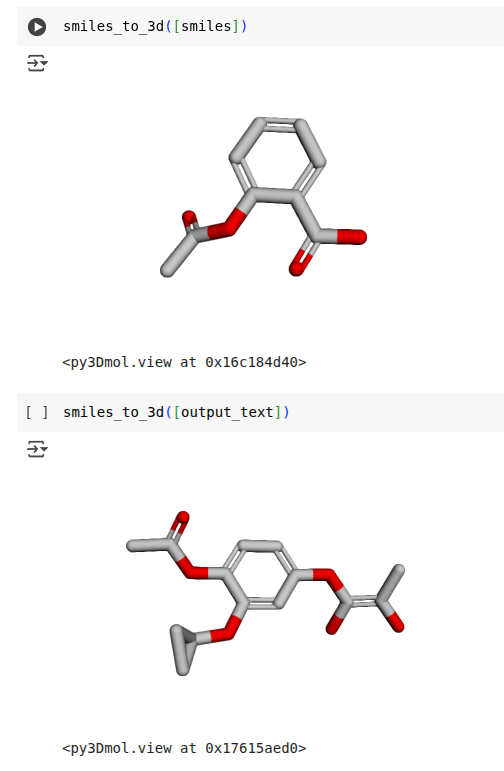
Dans l’exemple ci-contre issu du notebook associé au projet, en partant de la molécule du haut, l’outil génère la molécule du bas car il ne sait pas combien d’atomes il doit ajouter.
Pour résoudre en partie ce problème, il faudrait agrandir le fingerprint en y ajoutant une partie liée au nombre d’atomes de chaque type. Pour cela, il faudrait utiliser par exemple 50 bits pour indiquer le nombre les carbones aliphatiques, 50 pour le nombre de carbones aromatique, 25 pour les atomes d’oxygène sp3, 25 pour les atomes d’oxygène sp2, 25 pour les atomes d’azote sp3, 25 pour les atomes d’azote sp2, 25 pour les atomes d’azote sp, 10 pour les atomes de S sp3, 10 pour les atomes de S spd, 10 pour les atome de P spd, 10 pour chaque atome d’halogène. A chaque la position d’un 1 indiquera le nombre d’un type d’atome. Ce vecteur pourrait être également hashé.
En ajoutant une partie dédiée au nombre d’atomes, il est possible d’ajouter l’information manquante pour que le système puisse apprendre.
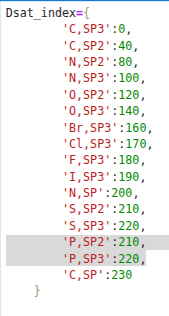
Position du début de codage pour le nombre d’atome d’un type donné.
