Analyse des résultats expérimentaux dans la Chembl
Introduction
La base de donnée nommée Chembl est vraiment très intéressante pour essayer de comprendre les mécanismes en jeu lors de l’interaction entre un ligand et une protéine. En effet, sur le site de la Chembl, on trouve un lien vers les résultats expérimentaux liés aux cibles biologiques. En bas à gauche sur l’image ci-dessous.
En triant les cibles par nombre de molécules (compounds) testées sur une cible, on constate que certaines cibles ont été vraiment très étudiées:
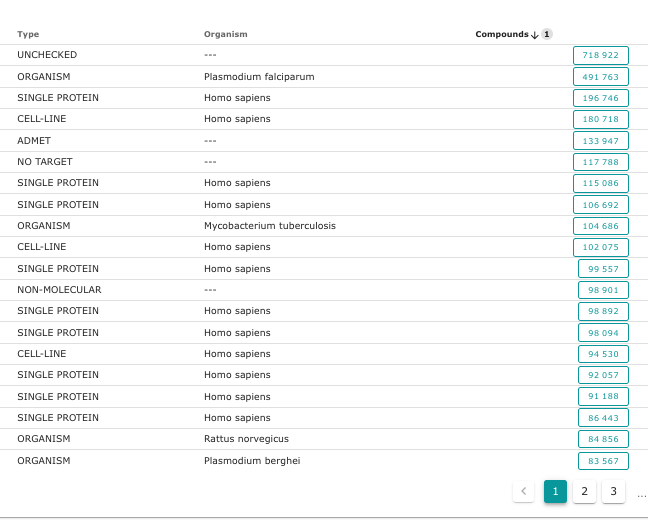
Ce qui nous intéresse en premier lieu, ce sont les résultats expérimentaux sur des “single proteins”, des protéines prises seules.
Pour commence une première analyse, j’ai choisi une protéine avec quelques milliers de molécules testées sur celle-ci, la CHEMBL3880, la “Heat shock protein HSP 90-alpha” avec 3019 molécules dont l’identifiant UniProt est P07900.
Un filtre sur le nombre maximum d’atomes (naat_max=30) a été appliquée et seules les molécules avec uniquement des atomes de chimie organiques ont été conservées soit 1855 molécules.
Le docking de ces molécules sur le site d’un inhibiteur dans le pdb 2YES donne le résultat suivant:
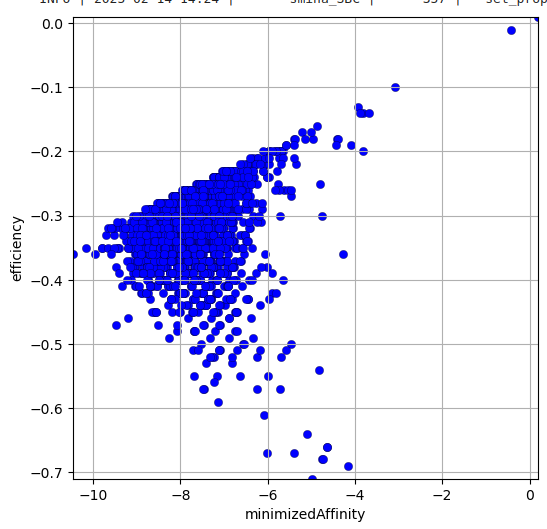
Molécules ayant une valeur de “‘minimizedAffinity” inférieure à -9,75:
 IC50 = 1700 nM IC50 = 1700 nM | 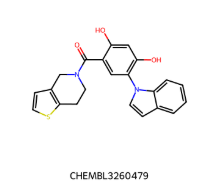 IC50 = 330 nM IC50 = 330 nM |
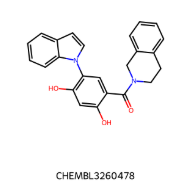 IC50=130 nM IC50=130 nM | 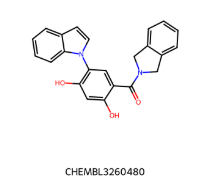 IC50 = 38 nM IC50 = 38 nM |
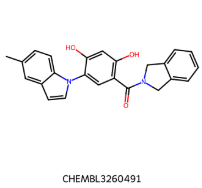 IC50=38 nM IC50=38 nM |
La molécules ayant la meilleure activité est la suivante:
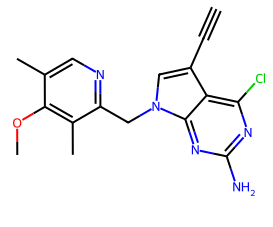 CHEMBL2205245 avec un IC50 de 1nM mais minimizedAffinity: -7.5 et efficiency: -0.31 CHEMBL2205245 avec un IC50 de 1nM mais minimizedAffinity: -7.5 et efficiency: -0.31 |
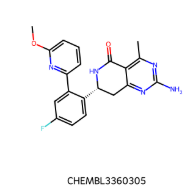
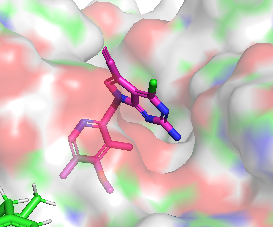
Différence de conformation entre CHEMBL3360305 et CHEMBL2205245
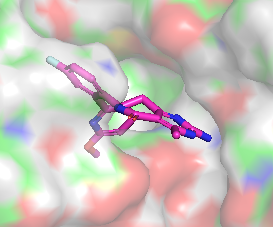 | 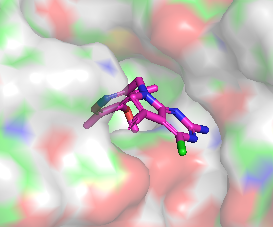 |
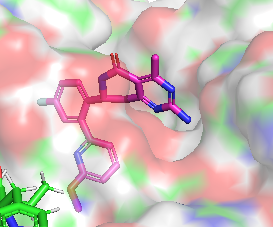 |  |
