Analyse des résultats expérimentaux dans la Chembl
Introduction
La base de donnée nommée Chembl est vraiment très intéressante pour essayer de comprendre les mécanismes en jeu lors de l’interaction entre un ligand et une protéine. En effet, sur le site de la Chembl, on trouve un lien vers les résultats expérimentaux liés aux cibles biologiques. En bas à gauche sur l’image ci-dessous.
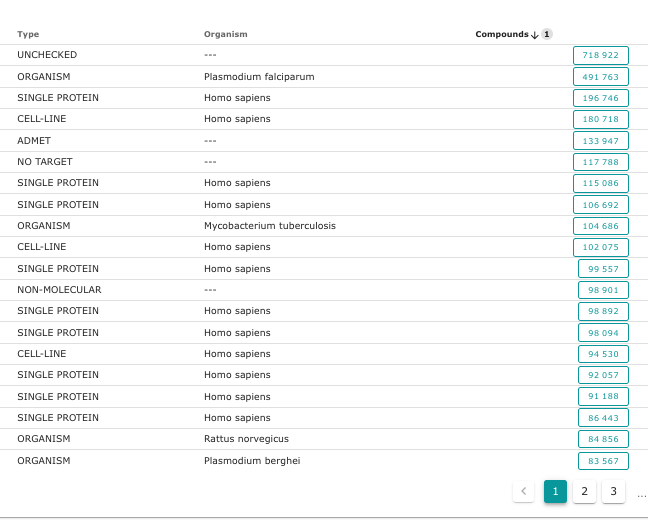
En triant les cibles par nombre de molécules (compounds) testées sur une cible, on constate que certaines cibles ont été vraiment très étudiées:
Ce qui nous intéresse en premier lieu, ce sont les résultats expérimentaux sur des “single proteins”, des protéines prises seules.
Pour commence une première analyse, j’ai choisi une protéine avec quelques milliers de molécules testées sur celle-ci, la CHEMBL3880, la “Heat shock protein HSP 90-alpha” avec 3019 molécules dont l’identifiant UniProt est P07900.
Un filtre sur le nombre maximum d’atomes (naat_max=30) a été appliquée et seules les molécules avec uniquement des atomes de chimie organiques ont été conservées soit 1855 molécules.
Le docking de ces molécules sur le site d’un inhibiteur dans le pdb 2YES donne le résultat suivant:
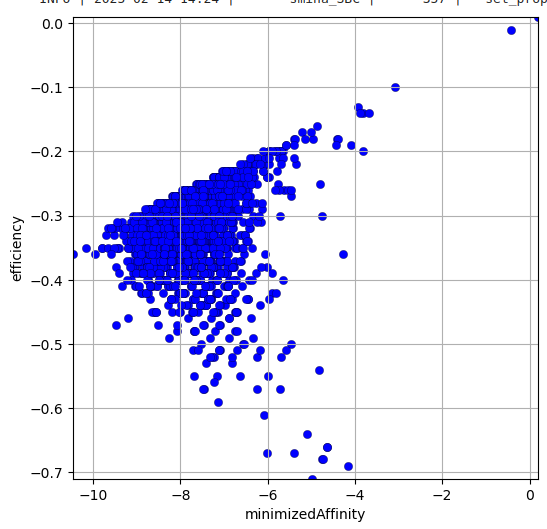
Molécules ayant une valeur de “‘minimizedAffinity” inférieure à -9,75:
 IC50 = 1700 nM IC50 = 1700 nM | 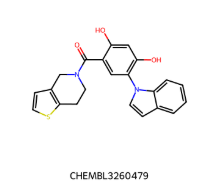 IC50 = 330 nM IC50 = 330 nM |
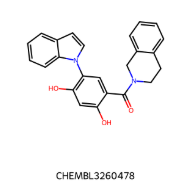 IC50=130 nM IC50=130 nM | 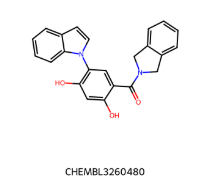 IC50 = 38 nM IC50 = 38 nM |
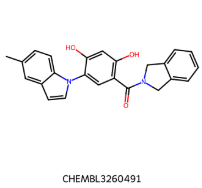 IC50=38 nM IC50=38 nM |
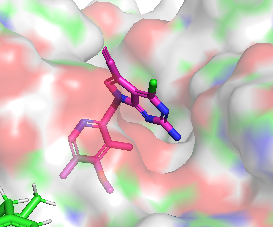
La molécules ayant la meilleure activité est la suivante:
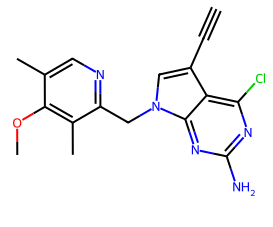 CHEMBL2205245 avec un IC50 de 1nM mais minimizedAffinity: -7.5 et efficiency: -0.31 CHEMBL2205245 avec un IC50 de 1nM mais minimizedAffinity: -7.5 et efficiency: -0.31 |
Différence de conformation entre CHEMBL3360305 et CHEMBL2205245
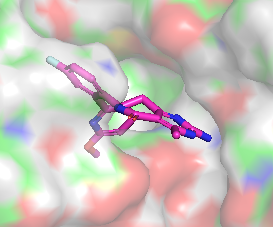 | 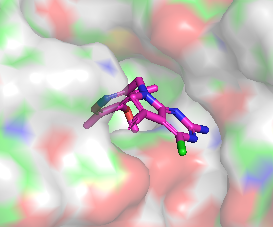 |
 |  |
Recherche de cas simples avec beaucoup de données pour tester les DNN
Question: Est-il possible de trouver des exemples d’applications des DNN où les données sont abondantes ou peuvent être générées rapidement ?
La spectrométrie de masse des peptides (enchaînement de moins de 30 acides aminés) pourrait être une possibilité. Dans la base de données MassIVE, on trouve plus de 40 M de spectres de masses de peptides!
Simplification de la recherche des pKa d’une molécule donnée.
Par Pascal KREZEL, ICOA, le 12/02/2025
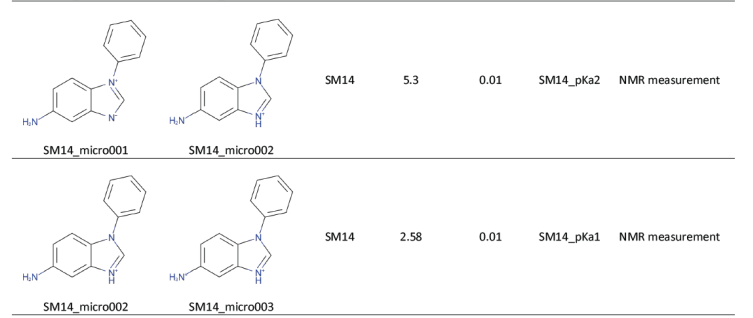
Les modèles actuels de prédiction de pKa cherchent à définir un pKa pour tous les sites d’une molécule. Or expérimentalement, une molécule possède moins de pKa que de fonctions acido-basiques. L’exemple suivant illustre ce problème. Dans SAMPL6 pKa Challenge, on trouve par exemple la molécule suivante avec 2 pKa.
Si on demande à molGpKa, une prédiction des pKa, sa réponse est la suivante:
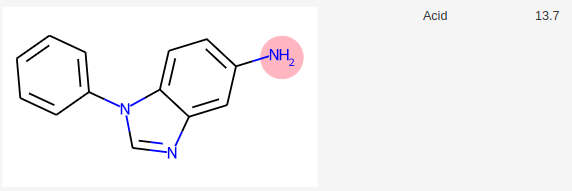
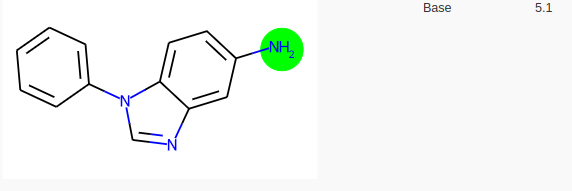
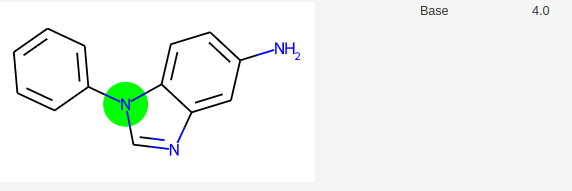
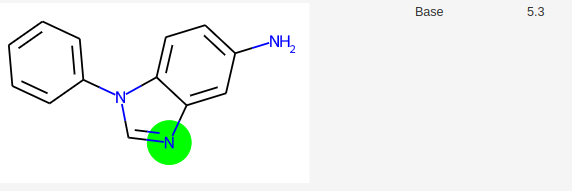
MolGpKa prédit donc 4 pKa.
Pour essayer de résoudre ce problème, nous proposons une nouvelle méthode passant par la prédiction d’une seule valeur, la charge moyenne à un pH donné.
Soit le cas extrême d’une molécule possédant 4 pKa dont il faut prédire les valeurs. Supposons qu’elle ait 2 fonctions acides et 2 fonctions basiques soit les équilibres suivants:
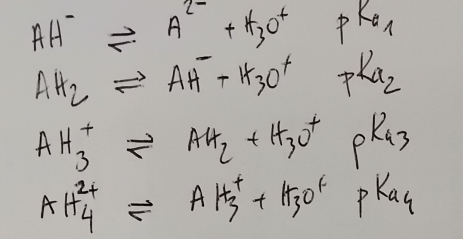
A pH fixé, on a les équations suivantes:
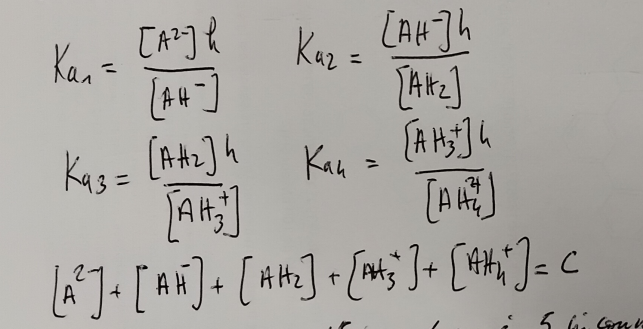
Ceci nous permet d’en déduire la charge moyenne à un pH donné:
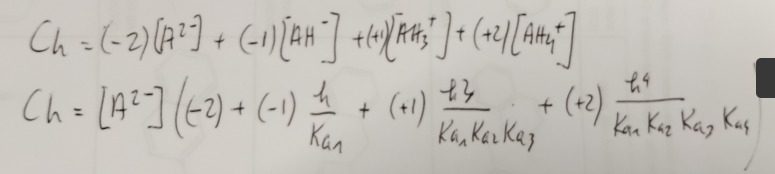
On en déduit une valeur de Ch dépendant uniquement de h et de C, la concentration totale.
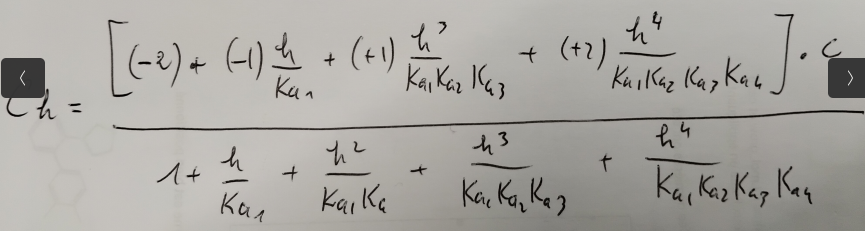
Finalement en fixant C à 1M, avec 4 valeurs de Ch pour différentes valeurs de pH, il est possible d’en déduire la valeur des différents Ka.
Si le système ne possède pas de solutions approximatives ou si les valeurs de Ka ne sont pas sont aberrantes, il faut en déduire que l’hypothèse des 2 fonctions acides et des 2 fonctions basiques était fausse et en prendre une autre.
Ainsi, en faisant au moins 4 modèles de prédiction de Ch pour différentes valeurs de pH, on doit être en mesure d’en déduire les différents pKa d’une molécule ayant au plus 4 pKa.
